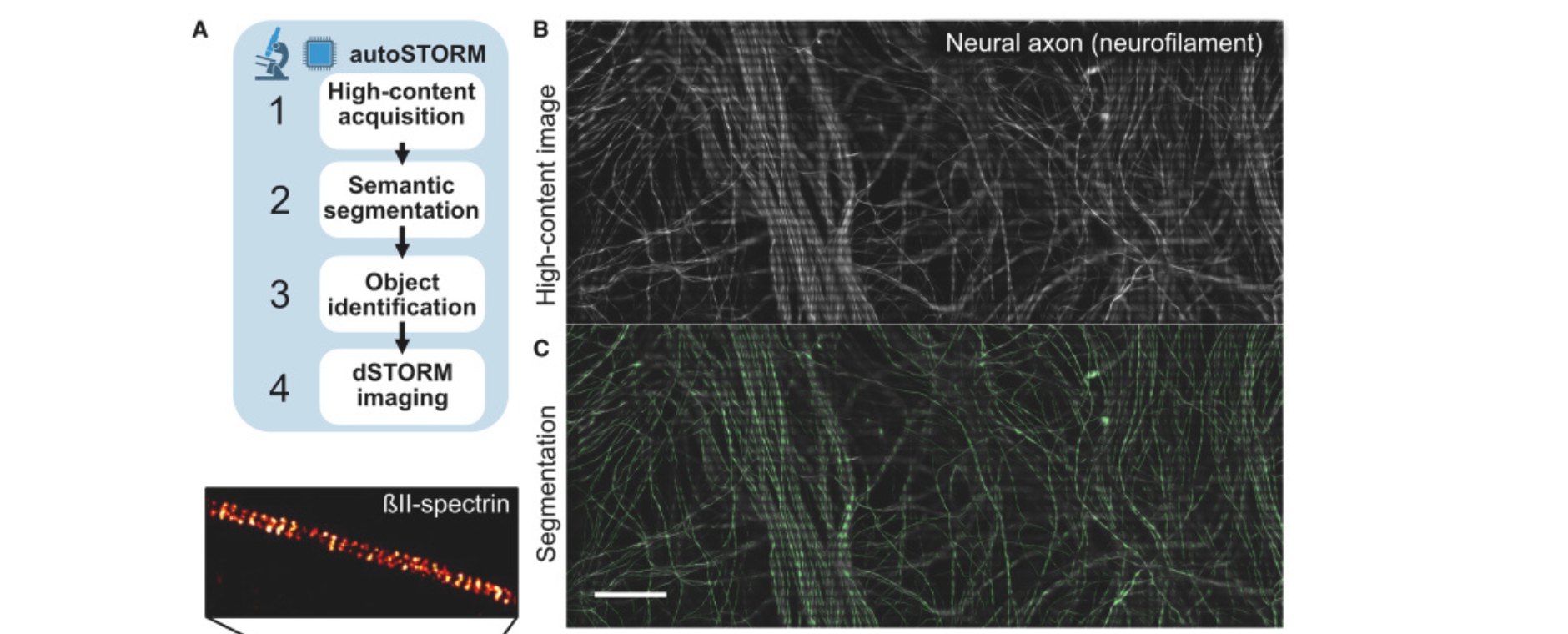
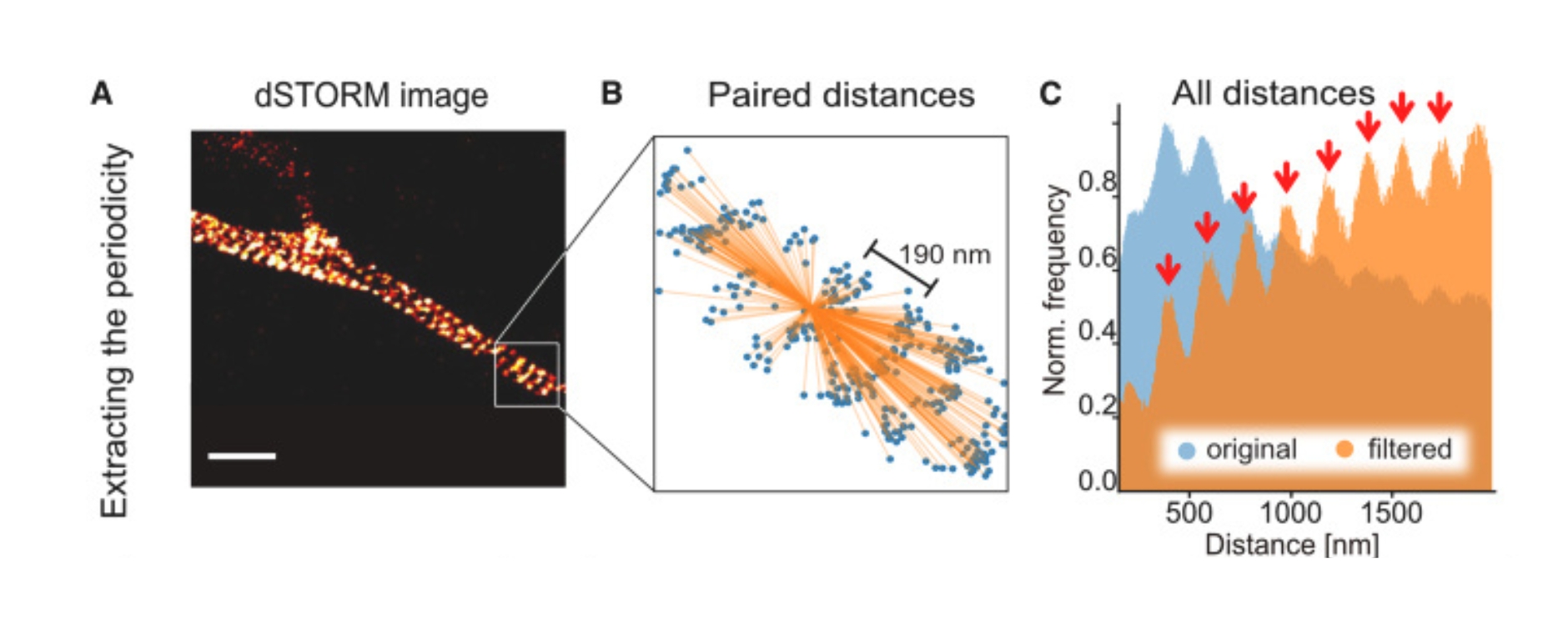
In dieser Studie präsentiert die Neurologie gemeinsam mit dem Rudolf-Virchow-Zentrum – Center für Integrative und Translationale Bildgebung (RVZ) ein skalierbares Open-Source-Software-Toolkit, das die Bilderfassung mit dSTORM automatisiert. Durch die Nutzung von Deep Learning zur Segmentierung kann das Toolkit Objekte in verschiedenen biomedizinischen Proben präzise identifizieren und gezielt anvisieren, selbst solche mit geringem Kontrast. Diese Automatisierung beschleunigt die Workflows dieser Bildgebung erheblich. Durch die Bereitstellung einer breit zugänglichen, benutzerfreundlichen Lösung können Forscherinnen und Forscher verschiedener Disziplinen die Leistungsfähigkeit der Super-Resolution-Mikroskopie nutzen, ohne eine umfangreiche Spezialausbildung zu benötigen.
Das eigenständige Programm, das in der Fachzeitschrift Biophysical Reports präsentiert wird und sogar den Titel erhalten hat, ermöglicht die zuverlässige Segmentierung biomedizinischer Bilder und übertrifft bestehende Lösungen. Integriert in die Bildgebungs-Pipeline verarbeitet es hochaufgelöste Daten in Minutenschnelle und reduziert so den manuellen Arbeitsaufwand. Anhand biologischer Beispiele wie Mikrotubuli in Zellkulturen und dem βII-Spektrin in Nervenfasern zeigen Janis T Linke und Katrin Heinze vom RVZ gemeinsam mit Luise Appeltshauser und Kathrin Doppler aus der Neurologie, dass der Ansatz die superauflösende Bildgebung schneller, robuster und benutzerfreundlicher macht, auch für Mikroskopie-Laien. Dies erweitert die Anwendungsmöglichkeiten in der Biomedizin, einschließlich Hochdurchsatz-Experimenten.
Janis T Linke, Luise Appeltshauser, Kathrin Doppler, Katrin G Heinze. Deep learning-driven automated high-content dSTORM imaging with a scalable open-source toolkit. Biophys Rep (N Y). 2025 Jun 11;5(2):100201. https://doi.org/10.1016/j.bpr.2025.100201. Epub 2025 Feb 28. PMID: 40023500; PMCID: PMC11986538.